
- 首页
- >
- GENESEED
- >
以上帝视角,从全转录组测序结果挖掘“宝藏”RNA
RNA近年蝉联话题MVP
mRNA:席卷研究全域,技术应用先锋,前景无限!
miRNA:诺奖引爆,药物临床III期成功,热潮再起!
circRNA:突破迈入临床,稳居RNA研究“C”位!
lncRNA:稳居研究前沿,“隐藏宝藏”,潜力巨大!
RNA研究不迷路
全转录组测序分析描绘“全景寻宝地图”
全转录组测序可同时得到mRNA、lncRNA、circRNA、miRNA等多种RNA的数据,能“一举”得到多种RNA的表达及调控关系,提供更全面、系统的基因表达信息。(注:通常全转录组测序单文库指仅长链RNA建库,包括mRNA、lncRNA、circRNA等数据;双文库指长链和小RNA分别建库,即包括miRNA数据。)
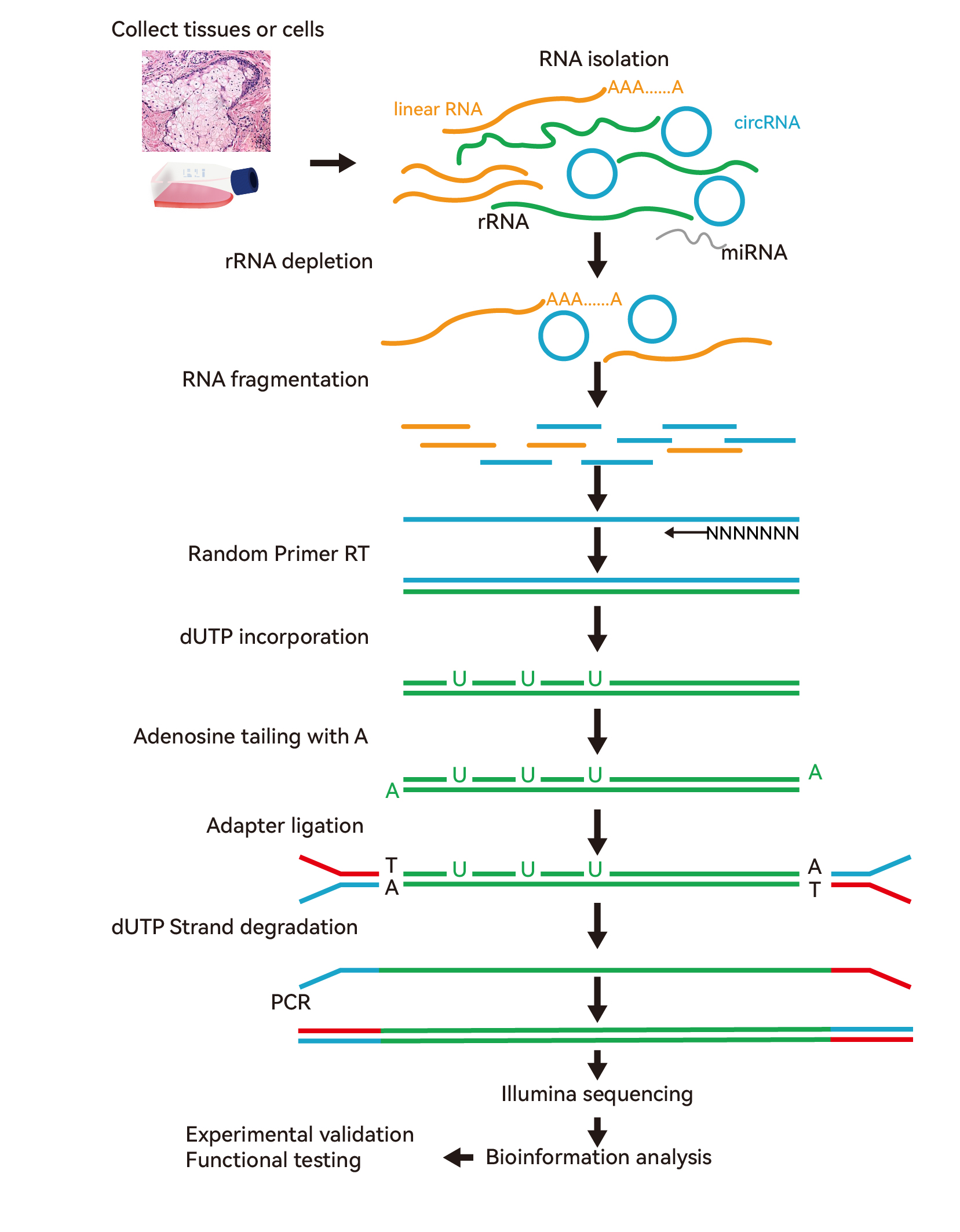
长链RNA测序文库构建流程
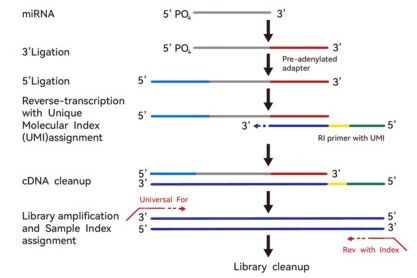
小RNA测序文库构建流程
从“全景地图”中快速捕捉关键“坐标”
差异表达分析——表型相关的“地标”
根据分组信息及过滤后的基因表达谱进行基因差异表达分析,可得到研究对象(实验组)中相比对照组差异表达的候选基因,如疾病、药物作用、环境变化、表型差异相关RNA。
差异表达分析输出的关键数据
Fold change(FC):基因表达变化的差异倍数,指示实验组与对照组之间基因表达水平的相对差异,反映基因表达上调或下调幅度。FC>1表示基因在实验组表达上调,FC<1表示基因在实验组表达下调。如FC=2表示实验组表达量是对照组的2倍;FC=0.5表示实验组表达量比对照组下调一半。
Log2FC:FC数值以2为底的对数转换,负数表示基因下调,正数表示基因上调。
P Value:统计检验中衡量统计学差异显著性的数值,指示数据差异的可信度,P值越小,差异的可信度越高。一般认为P Value<0.05表示统计学差异显著。
FDR:错误发现率,是统计学中用于多重假设检验的关键指标。基因表达分析中要同时检验成千上万个基因的表达差异。P Value会随着检验次数增加,误报次数也会增加。而FDR允许一定比例的误报,通过量化这个比例实现更灵活的错误控制。
Tn/Cn:实验组/对照组标准化后的相对表达量。
差异基因筛选
最高最显眼的很可能就与我们的研究目标最相近!在显著性差异表达基因表格中,对Fold Change和P Value分别进行主次排序,综合筛选表达差异倍数较大、差异显著、表达量合适并且感兴趣的候选基因,可以优先选择Fold Change > 2、P < 0.01的基因。
太阳成tyc33455cc全转录组测序可视化差异分析
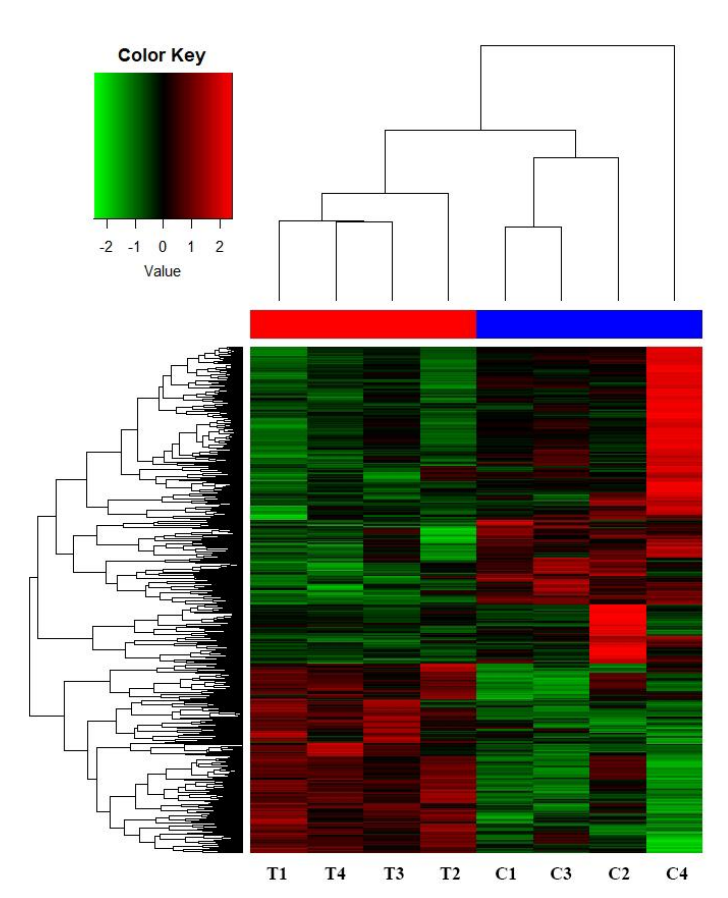
聚类热图
每列代表一个样本,每行代表一个基因,红色为表达上调,蓝色为表达下调。组内样本表达模式高度相似(颜色块整体一致),表明组内重复性好。组间样本颜色明显区分,表明两组基因表达差异显著 。
火山图
平行于X轴的虚线为设定的P value阈值(一般为0.05),虚线以上为差异显著的基因,这些基因数量越多,表明实验效果越明显。
平行于Y轴的虚线为设定的log2FC和-log2FC的阈值,分布越靠两侧的基因,表达差异程度越大。
顶端的点是核心差异基因,往往是通路关键节点或功能marker,可考虑用于后续验证。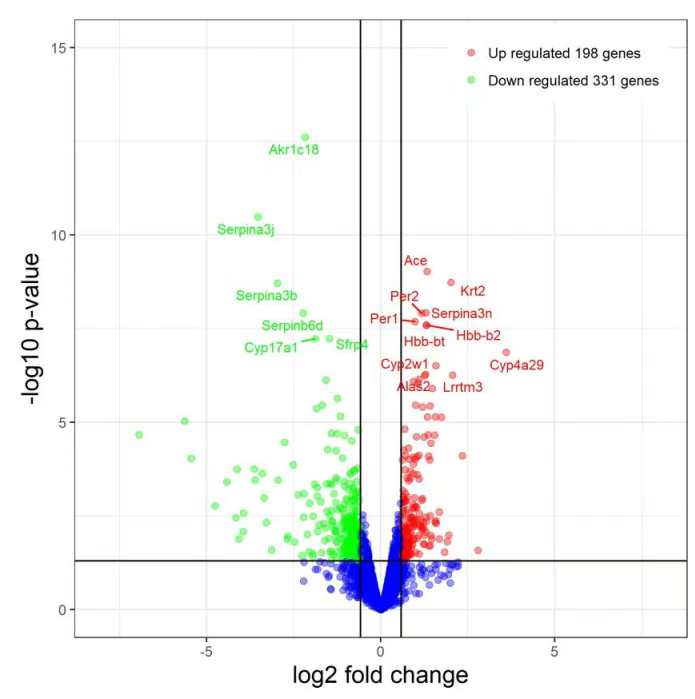
图5 体外合成的circRNA、线性RNA、Miravirsen和HCV RNA共转染到HuH-7.5细胞5天后,HCV NS3和core蛋白丰度。
通路富集分析——“地标”附近的“交通“概况
对差异表达基因进行功能注释和通路富集分析,可以:
● 表征实验处理影响最大和最针对的功能类型或信号通路;
● 进一步筛选实验处理影响最大的功能或信号通路相关的基因;
● 为分子机制验证提供基础。
太阳成tyc33455cc全转录组测序可视化通路富集分析
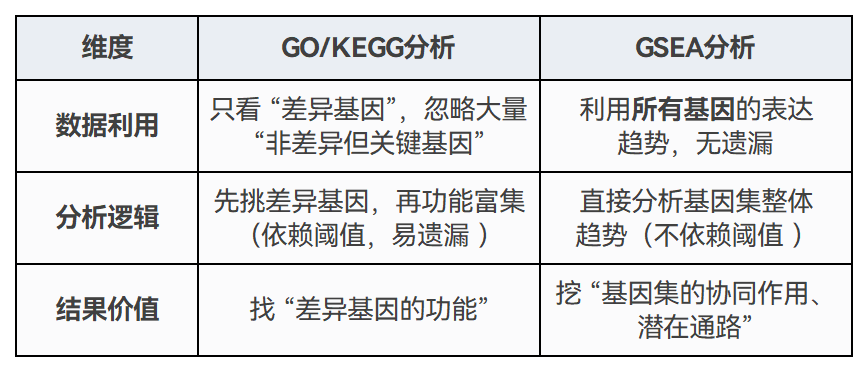
Gene Ontology(GO)分析
系统性注释物种基因及其产物属性,可分为分子功能,生物过程和细胞组成三个部分。柱越高,表明该功能中富集的差异基因越多,该功能受实验处理对该功能相关的基因针对性越强。
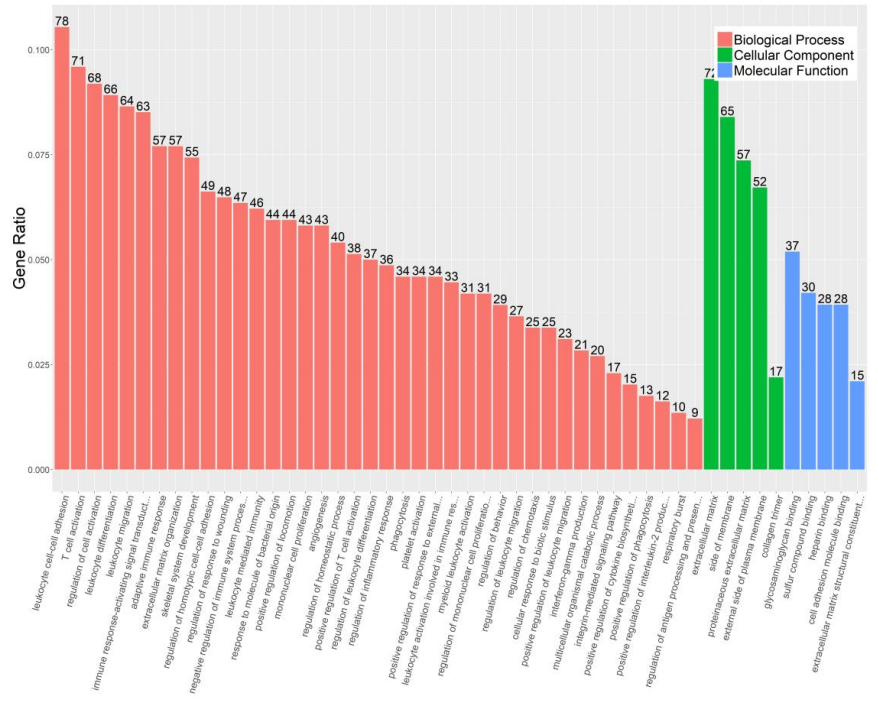
KEGG/Reactome通路富集分析
柱状图
通路显著性:色越蓝,-lgP越高,差异基因通路富集越显著,即实验处理对该通路影响越显著。如下图中的Cell cycle通路最高蓝,表明细胞周期通路富集最显著。
基因规模:Count越大,涉及的差异基因越多。如下图Cell cycle通路Count最大,可能是实验表型的核心通路。
通路优先级:按-lgP排序,前几位可能是最值得的通路 ,集中了差异基因和显著性。

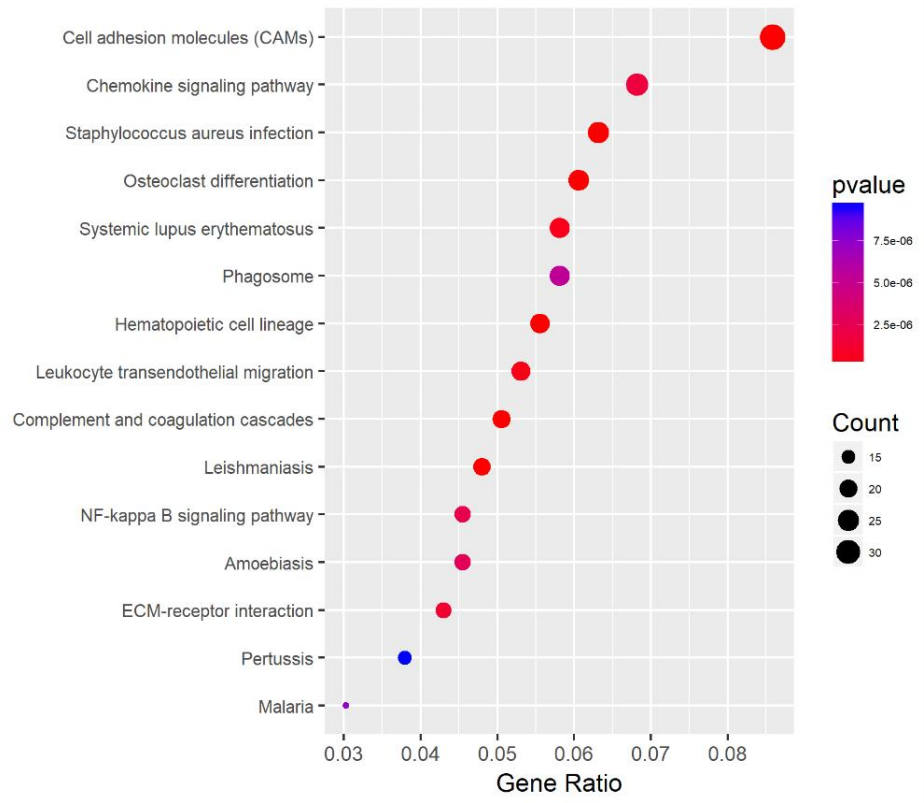
气泡图
通路关联性:Gene Ratio越大(靠右),该通路的差异基因占比越高,实验处理对该通路的针对性越强。
显著性与基因规模:气泡大(Count高 )、色蓝(-lgP高)、靠右(Gene Ratio高)的基因,是“三优”核心通路,可能是实验表型的主因。
通路分层:不同通路在Gene Ratio-lgP中的分布,能区分“基因多但显著性一般”和 “基因少但显著性高”的通路,辅助筛选研究重点。
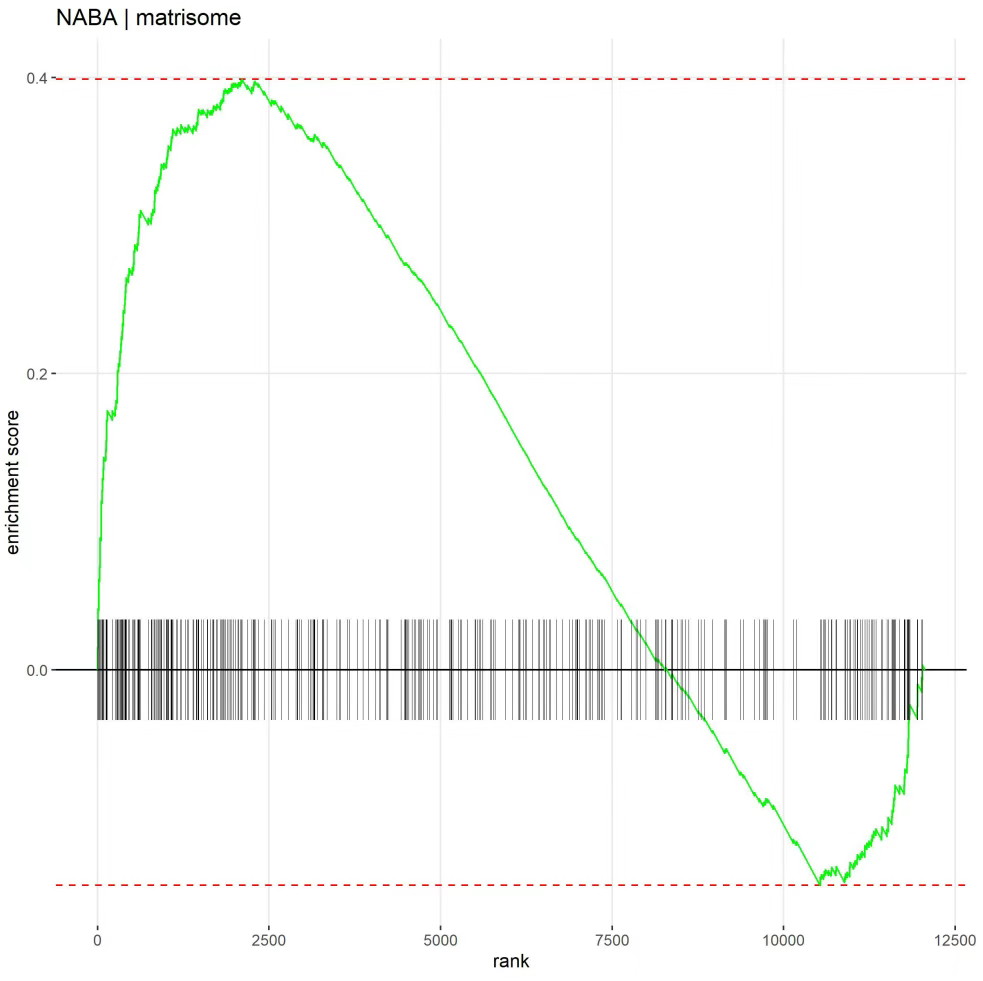
GSEA分析
不依赖于预先筛选差异表达基因,而直接对所有基因根据表达水平进行排序,然后判断基因集在排序后的基因列表中的分布情况,评估基因集是否在特定表型或条件下显著富集 。
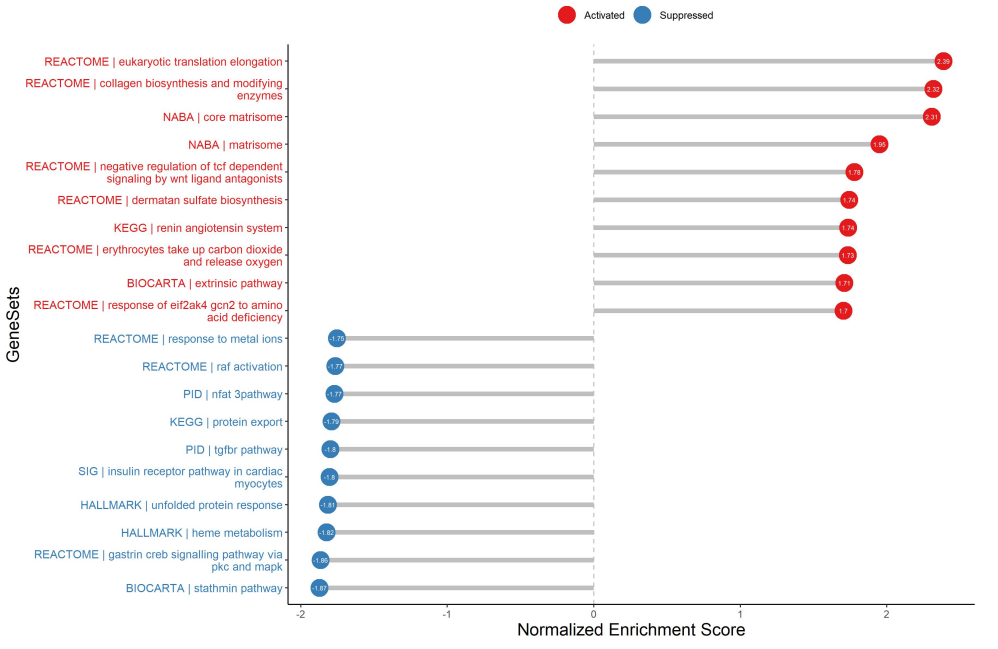
棒棒糖图
点的位置对应基因集的归一化富集分数,红色点代表在特定条件下基因集是激活状态,蓝色点代表抑制状态(红色点越靠右,富集越显著;蓝色点越靠左,抑制越显著)。
富集曲线图
上半部分曲线:表示运行富集分数。曲线的波动反映了基因集内的基因在排序后的基因列表中的分布情况。曲线上升表示基因集内的基因在当前位置的基因列表中出现频率较高;曲线下降则相反。曲线的最高点对应的富集分数就是该基因集的富集分数(ES),用于评估基因集的富集程度。
下半部分条形图:黑色竖条表示基因集中的基因在排序后的基因列表中的位置。竖条越密集,表明基因集内的基因在基因列表中分布越集中。通常根据基因的表达情况(如上调或下调)进行颜色编码,展示基因集中基因的表达趋势。
调控/互作网络分析——“地标”附近的“交通网络”
根据得到转录组,分析基因间相互作用或相互调控的情况,为分子调控机制研究提供参考。
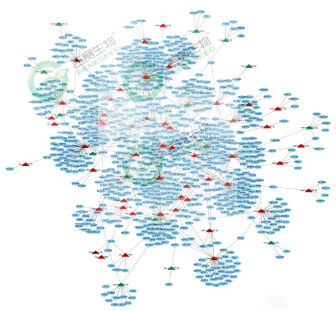
circRNA-lncRNA&circRNA-mRNA的共表达网络:指示协同作用的基因。
circRNA - miRNA互作网络图:预测差异表达circRNA互作的miRNA。
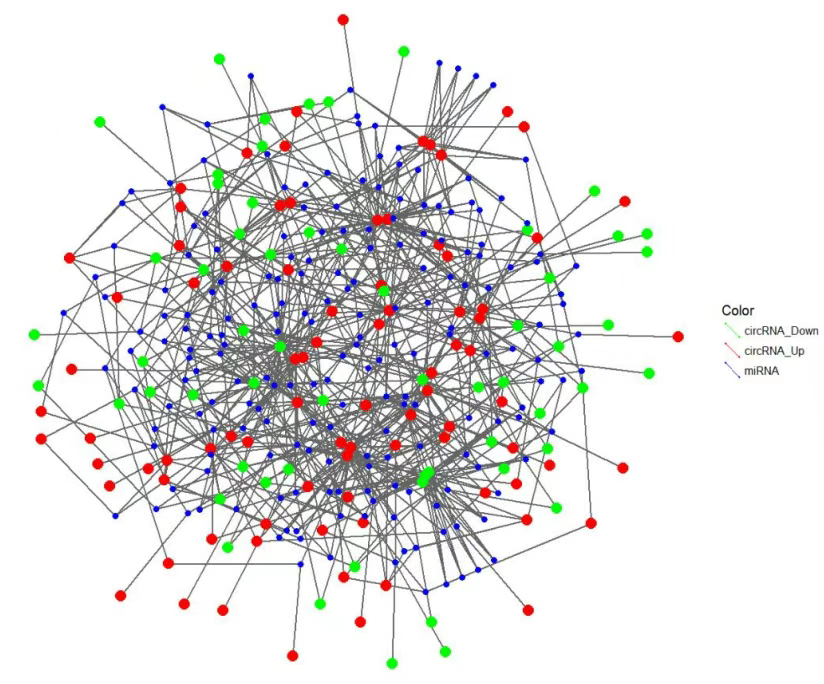
miRNA-基因调控网络:预测差异表达miRNA调控的基因。
参考文献
RNA近年蝉联话题MVP
mRNA:席卷研究全域,技术应用先锋,前景无限!
miRNA:诺奖引爆,药物临床III期成功,热潮再起!
circRNA:突破迈入临床,稳居RNA研究“C”位!
lncRNA:稳居研究前沿,“隐藏宝藏”,潜力巨大!
RNA研究不迷路
全转录组测序分析描绘“全景寻宝地图”
全转录组测序可同时得到mRNA、lncRNA、circRNA、miRNA等多种RNA的数据,能“一举”得到多种RNA的表达及调控关系,提供更全面、系统的基因表达信息。(注:通常全转录组测序单文库指仅长链RNA建库,包括mRNA、lncRNA、circRNA等数据;双文库指长链和小RNA分别建库,即包括miRNA数据。)

长链RNA测序文库构建流程

小RNA测序文库构建流程
从“全景地图”中快速捕捉关键“坐标”
差异表达分析——表型相关的“地标”
根据分组信息及过滤后的基因表达谱进行基因差异表达分析,可得到研究对象(实验组)中相比对照组差异表达的候选基因,如疾病、药物作用、环境变化、表型差异相关RNA。
差异表达分析输出的关键数据
Fold change(FC):基因表达变化的差异倍数,指示实验组与对照组之间基因表达水平的相对差异,反映基因表达上调或下调幅度。FC>1表示基因在实验组表达上调,FC<1表示基因在实验组表达下调。如FC=2表示实验组表达量是对照组的2倍;FC=0.5表示实验组表达量比对照组下调一半。
Log2FC:FC数值以2为底的对数转换,负数表示基因下调,正数表示基因上调。
P Value:统计检验中衡量统计学差异显著性的数值,指示数据差异的可信度,P值越小,差异的可信度越高。一般认为P Value<0.05表示统计学差异显著。
FDR:错误发现率,是统计学中用于多重假设检验的关键指标。基因表达分析中要同时检验成千上万个基因的表达差异。P Value会随着检验次数增加,误报次数也会增加。而FDR允许一定比例的误报,通过量化这个比例实现更灵活的错误控制。
Tn/Cn:实验组/对照组标准化后的相对表达量。
差异基因筛选
最高最显眼的很可能就与我们的研究目标最相近!在显著性差异表达基因表格中,对Fold Change和P Value分别进行主次排序,综合筛选表达差异倍数较大、差异显著、表达量合适并且感兴趣的候选基因,可以优先选择Fold Change > 2、P < 0.01的基因。
太阳成tyc33455cc全转录组测序可视化差异分析
聚类热图
每列代表一个样本,每行代表一个基因,红色为表达上调,蓝色为表达下调。组内样本表达模式高度相似(颜色块整体一致),表明组内重复性好。组间样本颜色明显区分,表明两组基因表达差异显著 。

火山图
平行于X轴的虚线为设定的P value阈值(一般为0.05),虚线以上为差异显著的基因,这些基因数量越多,表明实验效果越明显。
平行于Y轴的虚线为设定的log2FC和-log2FC的阈值,分布越靠两侧的基因,表达差异程度越大。
顶端的点是核心差异基因,往往是通路关键节点或功能marker,可考虑用于后续验证。
图5 体外合成的circRNA、线性RNA、Miravirsen和HCV RNA共转染到HuH-7.5细胞5天后,HCV NS3和core蛋白丰度。
通路富集分析——“地标”附近的“交通“概况
对差异表达基因进行功能注释和通路富集分析,可以:
● 表征实验处理影响最大和最针对的功能类型或信号通路;
● 进一步筛选实验处理影响最大的功能或信号通路相关的基因;
● 为分子机制验证提供基础。
太阳成tyc33455cc全转录组测序可视化通路富集分析
Gene Ontology(GO)分析
系统性注释物种基因及其产物属性,可分为分子功能,生物过程和细胞组成三个部分。柱越高,表明该功能中富集的差异基因越多,该功能受实验处理对该功能相关的基因针对性越强。

KEGG/Reactome通路富集分析
柱状图
通路显著性:色越蓝,-lgP越高,差异基因通路富集越显著,即实验处理对该通路影响越显著。如下图中的Cell cycle通路最高蓝,表明细胞周期通路富集最显著。
基因规模:Count越大,涉及的差异基因越多。如下图Cell cycle通路Count最大,可能是实验表型的核心通路。
通路优先级:按-lgP排序,前几位可能是最值得的通路 ,集中了差异基因和显著性。

气泡图
通路关联性:Gene Ratio越大(靠右),该通路的差异基因占比越高,实验处理对该通路的针对性越强。
显著性与基因规模:气泡大(Count高 )、色蓝(-lgP高)、靠右(Gene Ratio高)的基因,是“三优”核心通路,可能是实验表型的主因。
通路分层:不同通路在Gene Ratio-lgP中的分布,能区分“基因多但显著性一般”和 “基因少但显著性高”的通路,辅助筛选研究重点。

GSEA分析
不依赖于预先筛选差异表达基因,而直接对所有基因根据表达水平进行排序,然后判断基因集在排序后的基因列表中的分布情况,评估基因集是否在特定表型或条件下显著富集 。

棒棒糖图
点的位置对应基因集的归一化富集分数,红色点代表在特定条件下基因集是激活状态,蓝色点代表抑制状态(红色点越靠右,富集越显著;蓝色点越靠左,抑制越显著)。

富集曲线图
上半部分曲线:表示运行富集分数。曲线的波动反映了基因集内的基因在排序后的基因列表中的分布情况。曲线上升表示基因集内的基因在当前位置的基因列表中出现频率较高;曲线下降则相反。曲线的最高点对应的富集分数就是该基因集的富集分数(ES),用于评估基因集的富集程度。
下半部分条形图:黑色竖条表示基因集中的基因在排序后的基因列表中的位置。竖条越密集,表明基因集内的基因在基因列表中分布越集中。通常根据基因的表达情况(如上调或下调)进行颜色编码,展示基因集中基因的表达趋势。

调控/互作网络分析——“地标”附近的“交通网络”
根据得到转录组,分析基因间相互作用或相互调控的情况,为分子调控机制研究提供参考。
circRNA-lncRNA&circRNA-mRNA的共表达网络:指示协同作用的基因。
circRNA - miRNA互作网络图:预测差异表达circRNA互作的miRNA。

miRNA-基因调控网络:预测差异表达miRNA调控的基因。

参考文献

 购物车
购物车


 广州市黄埔区开源大道11号科技企业加速器A区6栋2楼
广州市黄埔区开源大道11号科技企业加速器A区6栋2楼


 geneseed@geneseed.com.cn
geneseed@geneseed.com.cn